(通讯员 易鸣)数学与物理学院“智能生物计算与数据挖掘”科研团队围绕生物医学和健康信息重要科学问题、瞄准新型计算和高效算法,近期取得系列进展,成果接连发表在《IEEE Journal of Biomedical and Health Informatics》、《PLOS Computational Biology》及《Chaos》等国际著名期刊。
多组学数据整合对癌症患者分类与生物标志物识别至关重要。尽管这种整合展现出巨大潜力,但其发展亟需建立精密的方法学框架。现有方法在同时满足组学特异性特征提取与跨组学关联建模需求方面仍存在显著提升空间。研究团队提出一种基于改进自编码器与注意力机制的深度学习框架“MOAEAM”以解决该问题。研究中提出了新颖的复合损失函数,有助于提取组学特异性特征,同时基于多组学整合模块通过捕捉跨组学信息共同提升分类性能。在构建组学自编码器时,为每个自编码器都结合特定的分类器,并采用了新颖的复合损失函数来优化分类性能,同时保留原始数据的结构特征。自编码器对所有组学数据进行降维处理后,通过注意力机制增强嵌入信息,然后对增强后的信息进行整合,整合后的结果包含组间和组内信息。在多组癌症数据集上的系统性评估表明,MOAEAM相较当前主流多组学整合方法能持续获得更优的分类性能。消融实验揭示,改进自编码器中引入的辅助分类器对性能提升起关键作用。通过模型计算的特征重要性评分可识别具有潜在临床意义的生物标志物,这些标志物已通过文献分析和富集分析得到进一步验证。该研究成果近日发表于《IEEE Journal of Biomedical and Health Informatics》,题为:“MOAEAM: Multi-omics data integration with autoencoder and attention mechanisms for cancer patient classification and biomarker identification”。 数学与物理学院2023级应用统计硕士生赵舒敏为该文的第一作者,易鸣教授为通讯作者。
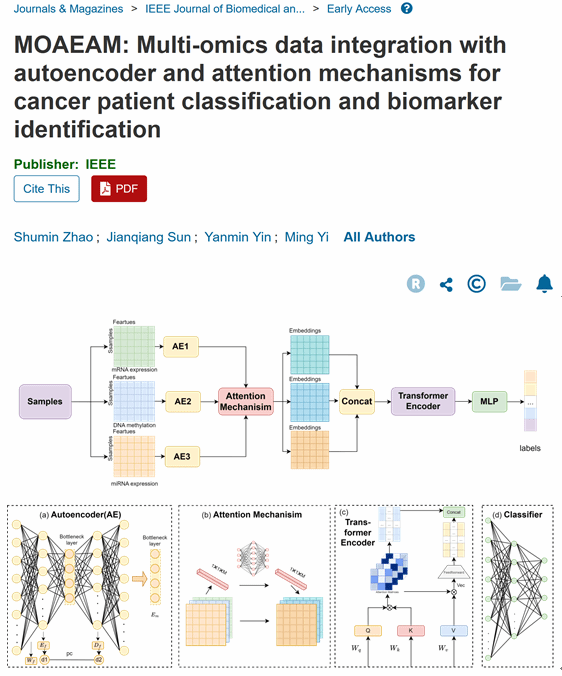
图1. MOAEAM算法流程图
单细胞RNA测序(scRNA-seq)为揭示细胞异质性提供了前所未有的机会,但从采样到测序过程中因技术因素导致的大量非生物学零值仍是数据分析中的核心难题。这些非生物零值掩盖了真实的基因表达模式,若与真正的生物零值混淆,可能严重扭曲下游分析结果。为解决这一挑战,研究团队提出了全新的计算框架D3Impute(Dropout-aware Discrimination, Distribution-aware Modeling, and Density-guided Imputation)。该框架的三大创新点包括:(1)分布感知的预处理去噪器通过建模基因表达分布的统计特性,自适应去除技术噪声并保留真实的生物学差异;(2)Dropout感知的判别器在双网络架构中整合了细胞-基因关系建模,将来自单细胞RNA测序(scRNA-seq)的细胞-细胞交互网络与来自群体RNA测序(bulk RNA-seq)的基因-基因交互网络进行协同结合,能够精准区分生物零与非生物零值;(3)密度感知的插补引擎在共享最近邻图中动态调节权重,选择性重建dropout值,保持局部异质性与全局结构。在实验验证中,研究团队首先进行了不同缺失率(60%、70%和80%)下的模拟实验,结果显示D3Impute 在高缺失条件下依然保持稳健的恢复能力。随后通过模块有效性验证实验,系统评估了三大核心模块的贡献,发现判别器与插补引擎的协同机制在提升零值识别与恢复准确性方面发挥了关键作用。最后在六组多样化数据集上与12种主流方法的系统性对比中,D3Impute在细胞聚类、轨迹推断和差异表达分析等关键下游任务中均表现出持续且显著的提升。综合分析显示,该方法不仅在不同数据质量条件下具备稳健性,还为研究者提供了明确的应用指南。这一成果为单细胞数据分析提供了更精准的工具,并为处理零膨胀数据建立了可推广的计算框架。该研究成果近日发表于《PLOS Computational Biology》,题为:“D3Impute: Dropout-aware discrimination, distribution-aware modeling, and density-guided imputation for scRNA-seq data”。 数学与物理学院2023级博士生黄斯怡为该文的第一作者,朱媛副教授为通讯作者。
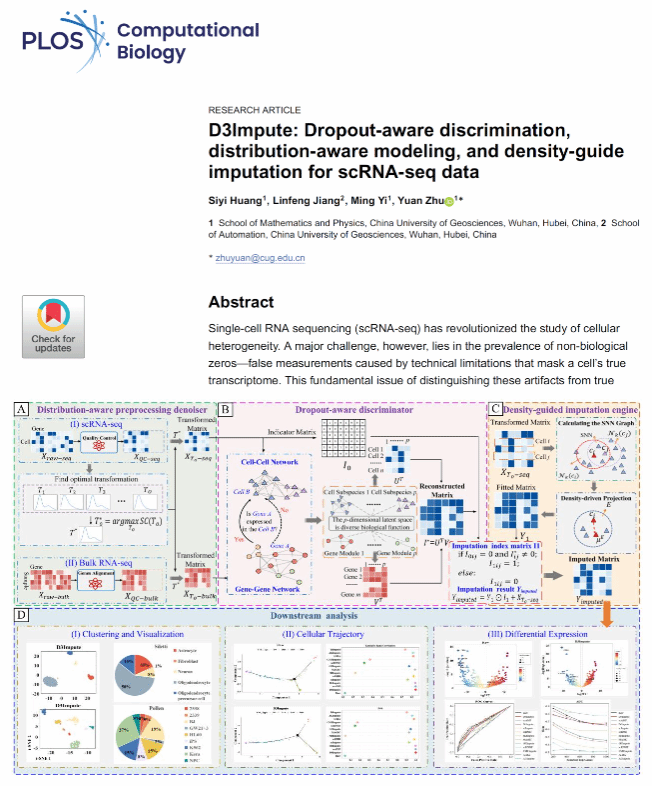
图2. D3Impute算法流程图
“智能生物计算与数据挖掘”科研团队陈小丽副教授指导数学与物理学院2023级数学硕士生柳江燕,聚焦基因调控随机系统逃逸问题的求解难点,创新引入深度学习方法构建求解模型,有效提升了复杂随机系统逃逸问题的求解效率,为该领域数值求解提供了新的技术思路;鹿露露副教授指导数学与物理学院2023级数学学硕程姗姗,瞄准神经系统中弱信号放大问题,基于可计算建模和数值模拟,发现了振子异质性诱导信号放大的新机制。以上系列成果近期发表于《Chaos》,见Chaos 35, 091107 (2025)、Chaos 35, 053150 (2025)、Chaos 35, 093136 (2025)。
IEEE Journal of Biomedical and Health Informatics是计算机科学和信息系统领域的领衔期刊之一,主要聚焦于交叉学科和生物医学与健康应用。该期刊为中科院认定的TOP期刊,JCR分区中在数学与计算生物学、医学等多个学科分类中均位列Q1分区,全球67种数学与计算生物学SCI收录期刊中排名第3。PLOS Computational Biology是计算生物学领域极具权威性的期刊,常年稳居JCR数学与计算生物学等学科Q1分区。《Chaos》期刊属于JCR数学学科Q1分区,刊登非线性动力学具有重要原创性的高水平研究论文。以上研究工作得到了国家自然科学基金(12371506、12305054))、广东省基础与应用基础研究基金(2025A1515010695)等项目资助。
(数学与物理学院来稿)